# Fiche d’identité : Osiris
# SYNTHÈSE :
Description et type de standard : OSIRIS (GrOupe inter-SIRIC sur le paRtage et l’Intégration des donnéeS clinico-biologiques en cancérologie) est un schéma de données théorique et un ensemble minimal de données qui permet de représenter les données cliniques et génomiques de patients atteints de cancer à travers une succession d'événements (antécédents, diagnostics, traitements, réactions, analyses, etc.). Il s’agit d’un standard français créé par l'Institut National du Cancer (INCa)[1].
Domaine d’application : Oncologie
Maturité / Utilisation : OSIRIS est un standard récent, dont la première version (v1) a été publiée en 2018 et la version actuelle (v1.1.05) est sortie en février 2019. À ce jour, il est utilisé par les institutions membres de l'initiative OSIRIS (Institut Curie, Institut Bergonié, Centre Léon Bérard, Institut du Cancer de Montpellier, Institut Paoli-Calmettes, Institut Gustave Roussy, CHU de Bordeaux, Hôpital Européen Georges Pompidou, Hôpital Saint Louis, Unicancer, voir l'élément « Adoption du standard » en partie 4. Valorisation).
# 1. Général
- Présentation :
- Pays d’origine : France
- Consortium d’origine : Institut National du Cancer (INCa)[2]
- Type de standard : Schéma de données et ensemble minimum de données
- Description :
- OSIRIS (GrOupe inter-SIRIC sur le paRtage et l’Intégration des donnéeS clinico-biologiques en cancérologie) est un schéma de données et un ensemble minimal de données cliniques et omiques lancé en 2015 par l’Institut National du Cancer (INCa) en France[3]. Il a pour objectif de permettre aux chercheurs et aux médecins de disposer dans le domaine de l'oncologie (1) de données homogènes (2) qui permettent de capter l'évolution de la maladie et en particulier la résistance aux interventions thérapeutiques et la toxicité. En particulier, il a été développé pour pouvoir analyser de manière conjointe les données issues de plusieurs essais cliniques menés dans les SIRICs (SItes de Recherche Intégrée sur le Cancer) en France.
- L'initiative OSIRIS propose de traiter le problème de l'hétérogénéité des données en prenant les quatre engagements suivants : (1) définir un ensemble minimal de données aussi restreint que possible, (2) atteindre un consensus national entre tous les acteurs intervenants dans la recherche sur le cancer, (3) utiliser des terminologies internationales aussi établies que possible, (4) définir des règles d'implémentation qui garantissent la cohérence de l'ensemble minimal de données dans les différentes institutions.
- Il existe plusieurs versions d’OSIRIS en cours d'élaboration mais nous concentrons dans la suite de la fiche sur OSIRIS Core qui est la seule version publiée en mai 2023[4].
- L'ensemble minimal de données est composé de 67 éléments cliniques et 65 éléments omiques.
- Organisme en charge : INCa et Unicancer[5].
Application :
- Domaine d'application en santé : Oncologie
- Principaux cas d'usage : Recherche clinique sur le cancer, essais cliniques, et en particulier dans le cadre du développement de la médecine de précision qui a conduit les SIRICs à mener des essais cliniques de profilage moléculaire[3:1].
- Illustration concrète, exemple d'utilisation sur un cas simple :
- Un test d'implémentation a été mené sur les données de 300 patients inclus dans 6 essais cliniques[3:2] :
- Dans les données cliniques :
- Une forte hétérogénéité entre les éléments communs de données (Common Data Elements) utilisés dans chaque essai clinique : certains concepts cliniques de l'ensemble minimal de données sont bien représentés (Patient, BiologicalSample, TumorPathologyEvent, Treatment, AdverseEvent, Drug) alors que d'autres sont très peu utilisés (FamilyCancerHistory, RelatedPathology)
- Des différences notables entre les terminologies utilisées : certains médecins utilisent des terminologies nationales (ex : la codification de l'ADICAP, Association pour le Développement de l'Informatique en Cytologie et Anatomie Pathologique) tandis que d'autres utilisent des terminologies internationales (ex : ICD-O-3, International Classification of Diseases for Oncology).
- Le processus de traitement de données suivant a été adopté : traduction des terminologies nationales vers les terminologies internationales, évaluation de la qualité des données, correction des erreurs et complétion des données manquantes
- Dans les données omiques :
- Tous les essais cliniques utilisent le séquençage de nouvelle génération (NGS : Next-generation sequencing)[3:3], ce qui permet une cohérence des concepts génomiques
- Données génomiques hétérogènes en raison de la variété des technologies NGS et des outils d'analyse (description des altérations génomiques hétérogènes ou partielle)
- Variabilité des métadonnées (interprétation clinique des variants, etc.).
- Dans les données cliniques :
- Pour permettre l'interopérabilité de l'ensemble minimal de données OSIRIS, il a été rendu compatible avec le standard HL7 FHIR : la partie génomique du schéma de données a été mappée aux ressources génomiques HL7 FHIR (voir l'élément « Description » plus haut).
- Un test d'implémentation a été mené sur les données de 300 patients inclus dans 6 essais cliniques[3:2] :
Données :
- Typologie de données concernées :
- L'ensemble minimal de données consiste en une liste de 132 éléments communs de données (Common Data Element), dont 67 correspondent à des données cliniques et 65 à des données omiques[6]. Les variables (ou Data Element ou DE, on parle aussi de « concepts ») peuvent correspondre aux types suivants : chaîne de caractères, date, nombre entier, nombre décimal.
- Les concepts représentés dans ces données sont les suivants :
- Concepts de données cliniques[7] : description du patient, consentement du patient, pathologies associées (autres que le cancer ; ex : diabète), antécédents carcinologiques du patient ou de ses apparentés, événement tumoral, analyse survenue au cours d'un événement tumoral, résultat pour un marqueur donné dans le cadre d'une analyse de biologie moléculaire survenue au cours d'un événement tumoral, traitement (chirurgie, chimiothérapie, radiothérapie, immunothérapie), événement indésirable, molécule administrée, échantillon biologique
- Concepts de données omiques : technologie utilisée, panel, analyse omique, altération génomique, méthode de validation de l'altération, variant, etc.
- Type de granularité :
- La granularité est au niveau d’un événement carcinologique : chaque variable (appelée « Data Element » ou DE) est liée à un concept TumorPathologyEvent (TPE), c'est-à-dire à une tumeur primaire, récidive locale ou récidive métastatique[3:4]. Cela permet de suivre l'évolution de la maladie dans le temps : OSIRIS est un modèle de données temporel fondé sur les événements (event-based temporal model).
- Utilisation dans plusieurs langues : Le schéma de données et l'ensemble minimal de données sont définis en anglais.
- Typologie de données concernées :
Disponibilité de la documentation d'implémentation :
Description technique du schéma de données :
- Le schéma de données OSIRIS est composé (1) d'un modèle de données cliniques et (2) d'un modèle de données omiques.
- Dans le modèle de données cliniques (voir Figure 1) chaque élément de donnée est lié à un concept de TumorPathologyEvent (tumeur primaire, récidive locale ou récidive métastatique). Le modèle a pour objectif de proposer une représentation événementielle de la maladie carcinologique de chaque patient du point de vue de ses données cliniques[7:1] (voir Figure 2) :
- Chaque patient est associé à des antécédents familiaux de cancer ainsi qu'à des pathologies liées
- Chaque patient a un ou plusieurs événements carcinologiques
- Pour chaque événement carcinologique, on associe :
- D'une part les données cliniques, à savoir :
- Le traitement
- La réponse au traitement
- Les effets indésirables à la suite du traitement
- D'autre part les données omiques, à savoir : les analyses réalisées sur un échantillon (image, omique, biologie, examen pathologique, ...). Ce dernier bloc de données fait le lien avec le modèle de données omiques.
- D'une part les données cliniques, à savoir :
- Pour chaque événement carcinologique, on associe :
- Le modèle couvre la description des tumeurs solides et des changements mineurs seront nécessaires pour couvrir les tumeurs hématologiques (tumeurs développées à partir de cellules du sang)[3:6].
- Dans le modèle de données cliniques (voir Figure 1) chaque élément de donnée est lié à un concept de TumorPathologyEvent (tumeur primaire, récidive locale ou récidive métastatique). Le modèle a pour objectif de proposer une représentation événementielle de la maladie carcinologique de chaque patient du point de vue de ses données cliniques[7:1] (voir Figure 2) :
- Le schéma de données OSIRIS est composé (1) d'un modèle de données cliniques et (2) d'un modèle de données omiques.
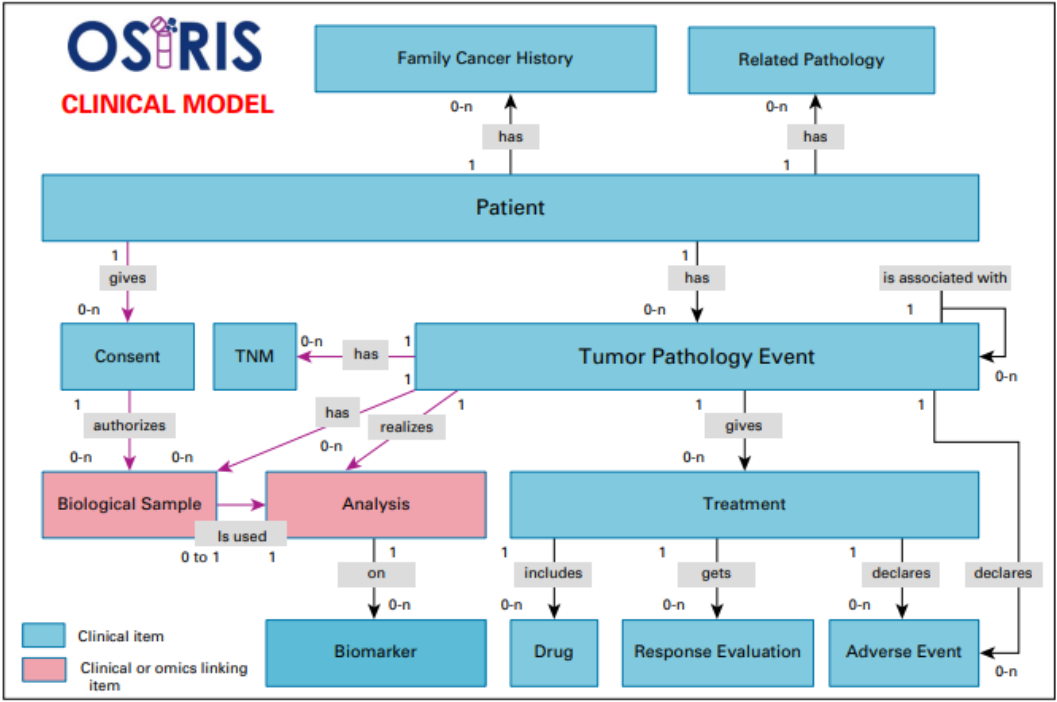
[Figure 1 : Modèle de données cliniques (OSIRIS), Source : Guérin et al. (2021)
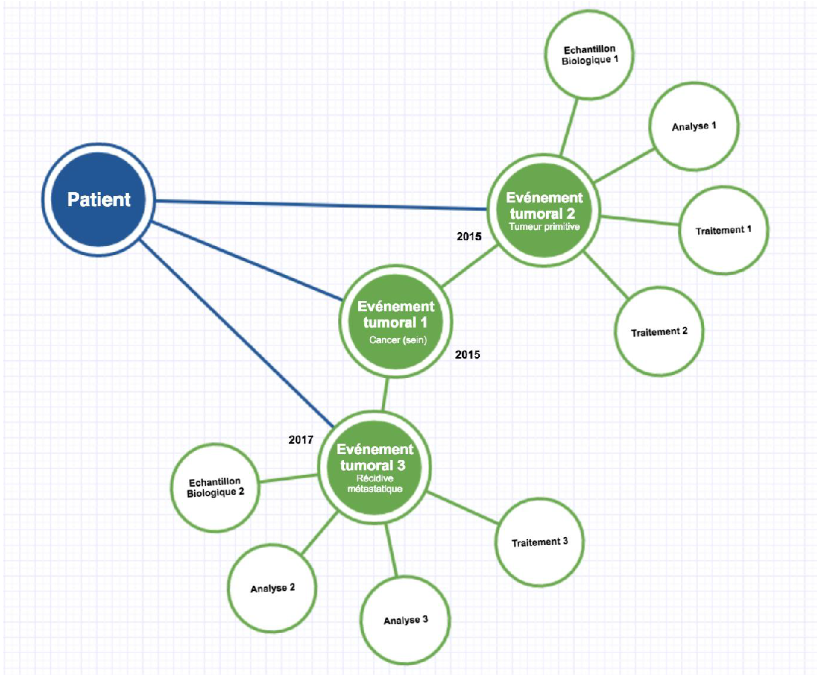
[Figure 2 : exemple des interactions entre les concepts du modèle clinique, Source : Description du Modèle de Données Cliniques (version 1.0, 2021)
- Dans le modèle de données omiques (voir Figure 3), tous les concepts sont liés au TumorPathologyEvent. Les concepts du modèle sont les suivants :
- Définition du contexte de l'analyse : technologie de séquençage, paramètres de l'analyse, etc.
- Niveau de confiance : des prédictions (validation) ou de l'annotation des variantes (annotation)
- Différents types d'altération sont pris en compte dans le modèle : nombre de copies, fusions, expression de gènes, mutations somatiques. La flexibilité du modèle permet d'ajouter de nouvelles données omiques dans le modèle (ex : épigénétique, protéomique).
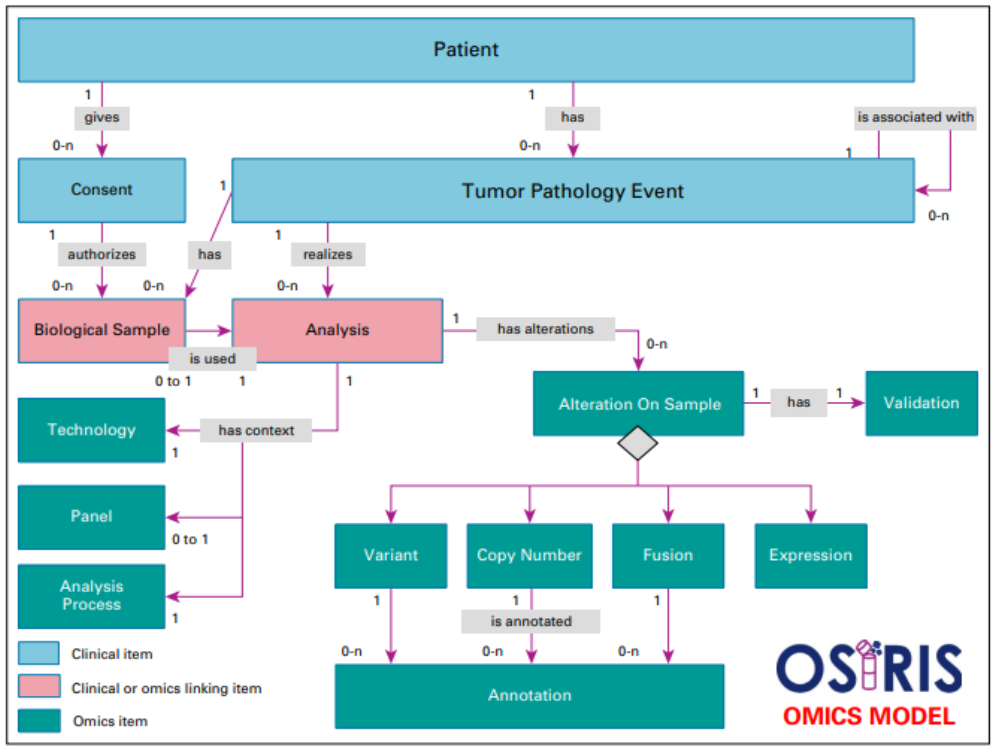
[Figure 3 : Modèle de données omiques (OSIRIS), Source : Guérin et al. (2021)
Les extensions du modèle contiennent d'autres composantes (voir l'élément « Flexibilité du standard, personnalisation » en partie 3. Technique et l'élément « Existence d’extensions certifiées » en partie 5. Utilisation).
Niveau de généralisation (facilité de remplissage des champs du standard) :
- Note : 0,6 / 1
- Cette note combine plusieurs sous-critères. Elle s’explique par :
- Le fait que les terminologies ne sont pas imposées, et en particulier il n’y a pas de terminologies locales imposées (voir l’élément « Flexibilité dans les choix des terminologies » en partie 3. Technique).
- L’absence de possibilité de personnalisation du standard malgré l’existence de quelques extensions (voir les éléments « Flexibilité du standard, personnalisation » en partie 3. Technique et « Existence d’extensions certifiées » en partie 5. Utilisation)
- L’absence de contraintes d’implémentation (voir l’élément « Contraintes d'implémentation » en partie 3. Technique)
- Une couverture moyenne des cas d’usage (voir l’élément « Principaux cas d'usage » en partie 1. Général)
- Une faible couverture des domaines d’application (voir l’élément « Domaine d'application en santé » en partie 1. Général)
- La facilité de remplissage des champs du standard dépend :
- De la disponibilité des données sources :
- Les tables du modèle OSIRIS font référence à des données spécifiques que l’on peut retrouver dans les bases de données des hôpitaux, des laboratoires, et d'autres établissements ayant une expertise en oncologie. Toutefois, en pratique plusieurs difficultés peuvent être rencontrées pour remplir ces champs :
- Dans les établissements de santé, ces données sont rarement stockées de manière centralisée, elles sont dispersées dans de nombreuses bases de données
- Selon les pratiques de l'établissement et des professionnels de santé, le niveau de complétude des champs ainsi que la profondeur de l'historique varient
- Les établissements de santé ont en général une vision limitée du parcours de soins, qui se limite au périmètre de leur établissement.
- Les tables du modèle OSIRIS font référence à des données spécifiques que l’on peut retrouver dans les bases de données des hôpitaux, des laboratoires, et d'autres établissements ayant une expertise en oncologie. Toutefois, en pratique plusieurs difficultés peuvent être rencontrées pour remplir ces champs :
- De la facilité à réaliser le mapping des données sources :
- Le modèle OSIRIS est extensible, modulaire et dispose d’extensions permettant de traiter divers types de données (imagerie, radiothérapie, etc., voir l'élément « Flexibilité du standard, personnalisation » en partie 3. Technique et l'élément « Existence d’extensions certifiées » en partie 5. Utilisation).
- De la disponibilité des données sources :
# 2. Gouvernance
Libre accès aux schémas de données : Oui
Modalités d'accès et distribution des solutions basées sur ce standard :
- Le modèle OSIRIS est en accès libre sur GitHub et n'est pas protégé par une licence[10].
Processus de prise de décision sur le standard :
- Le groupe de travail, mené par l'INCa, et contributeur au modèle de données initial est composé d'Unicancer, de plusieurs Centres de Lutte Contre le Cancer (CLCC)[11], de Centres Hospitaliers Universitaires (CHU)[12] et des huit SIRICs[13]. Les principaux CLCC ayant contribué sont l'Institut Curie, l'Institut Bergonié et le Centre Léon Bérard[14].
- La méthodologie d'élaboration du modèle OSIRIS a consisté à organiser des réunions hebdomadaires de plusieurs groupes nationaux (voir Figure 4) :
- Groupe SIRIC multidisciplinaire (composé d'oncologues, spécialistes d'informatique médicale, bio-informaticiens, épidémiologistes, bio-statisticiens, data managers, chercheurs, etc.) : sélection des données les plus pertinentes à inclure dans le modèle (Data Elements)
- Groupe scientifique (National Scientific Board composé d'oncologues, de chercheurs en recherche translationnelle en oncologie et de data protection officers) : examen de la conformité des données sélectionnées précédemment (Data Elements) au RGPD et à la réglementation CNIL
- Groupe technique (Technical Board composé de bio-informaticiens et data managers) : identification des terminologies nationales et internationales les plus pertinentes (ex : la CCAM pour les actes médicaux) et mapping des données (Data Elements) avec ces terminologies.
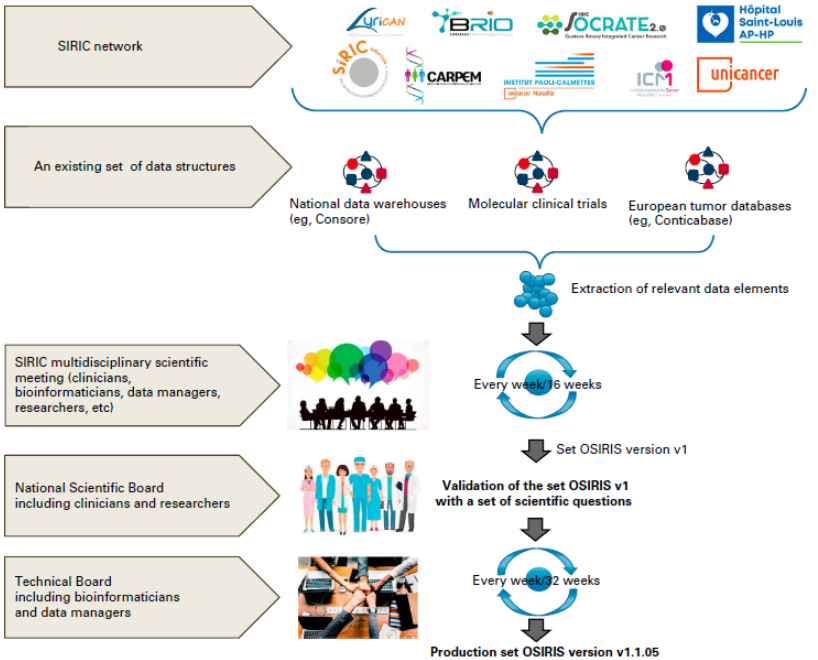
[Figure 4 : Méthodologie d'élaboration de la première version d'OSIRIS, Source : Guérin et al. (2021)
La procédure de contribution aux concepts (nouvelles extensions ou mise à jour de concepts existants) est décrite dans le GitHub[15] : la procédure est libre, les utilisateurs peuvent proposer des modifications (via une pull request) qui seront examinées par le groupe OSIRIS.
Des suggestions de modifications ou des bogues peuvent également être signalés dans le repository GitHub dédié[16]
Des questions peuvent être envoyées à l’adresse contact@siric-osiris.fr[15:1]
Maturité du standard :
- Fréquence de mise à jour :
- La première version du schéma de données OSIRIS (v1) est sortie en mai 2018
- Depuis cette date, 3 mises à jour de la v1 ont été publiées (v1.1.03, v1.1.04 et v1.1.05)[17] à la suite des échanges avec les groupes scientifique et technique (voir l'élément « Processus de prise de décision sur le standard » ci-dessus)
- En mai 2023, la dernière version est la v1.1.05 sortie en février 2019
- Maturité :
- Note : 0,4 / 1.
- Cette note combine plusieurs sous-critères. Elle s’explique par :
- La publication d’une version stable
- L’absence à date de mise à jour de la version principale
- Le jeune âge du standard
- Son utilisation dans le monde restreinte à l’échelle d’institutions (voir l’élément « Adoption du standard » en partie 4. Valorisation)
- L’absence d’adoption officielle par un ou plusieurs pays ou par une organisation de référence (voir l’élément « Adoption du standard » en partie 4. Valorisation)
- Fréquence de mise à jour :
Existence de financements pour standardisation : Non.
# 3. Technique
- Capacité de traduction vers un autre standard (intra types de standards) :
- OSIRIS est conçu comme un modèle de données dont l'un des objectifs principaux est d'être interopérable. Il définit l'ensemble minimal de données théorique, issu des données source (EHR, systèmes e-CRF tels que REDCap ou cdisc, ou entrepôt de données du type CONSORE), permettant d'analyser l'évolution de la maladie carcinologique. Il ne définit que la structure théorique, et non la structure physique des bases de données. Pour cela, des standards internationaux, qui contiennent une implémentation physique, sont utilisés dans une étape complémentaire, selon l'objectif recherché (voir Figure 5 ci-dessous), et les processus ETL associés sont développés en partenariat avec des sociétés tiers :
- Processus ETL OSIRIS-FHIR (travail réalisé en partenariat avec la société Arkhn)[18] : les Data Elements qui constituent l'ensemble minimal de données ont été associés aux ressources FHIR correspondantes (voir Figure 5 ci-dessous).
- Processus ETL OSIRIS-i2b2[19] : Pour rendre compatible OSIRIS et i2b2, un guide détaille les étapes de déploiement de l'instance i2b2/SHRINE et contient les fichiers permettant de charger les Data Elements OSIRIS
- Le consortium OSIRIS travaille actuellement à développer le processus ETL OSIRIS – OMOP-CDM[3:7] (voir Figure 5 ci-dessous)
- Le modèle OSIRIS a été comparé aux modèles mCODE et OMOP-CDM dans le Data Supplement n°3 (sur les caractéristiques générales, les composantes du modèle, les concepts disponibles et les terminologies utilisées)[6:3]
- OSIRIS est conçu comme un modèle de données dont l'un des objectifs principaux est d'être interopérable. Il définit l'ensemble minimal de données théorique, issu des données source (EHR, systèmes e-CRF tels que REDCap ou cdisc, ou entrepôt de données du type CONSORE), permettant d'analyser l'évolution de la maladie carcinologique. Il ne définit que la structure théorique, et non la structure physique des bases de données. Pour cela, des standards internationaux, qui contiennent une implémentation physique, sont utilisés dans une étape complémentaire, selon l'objectif recherché (voir Figure 5 ci-dessous), et les processus ETL associés sont développés en partenariat avec des sociétés tiers :
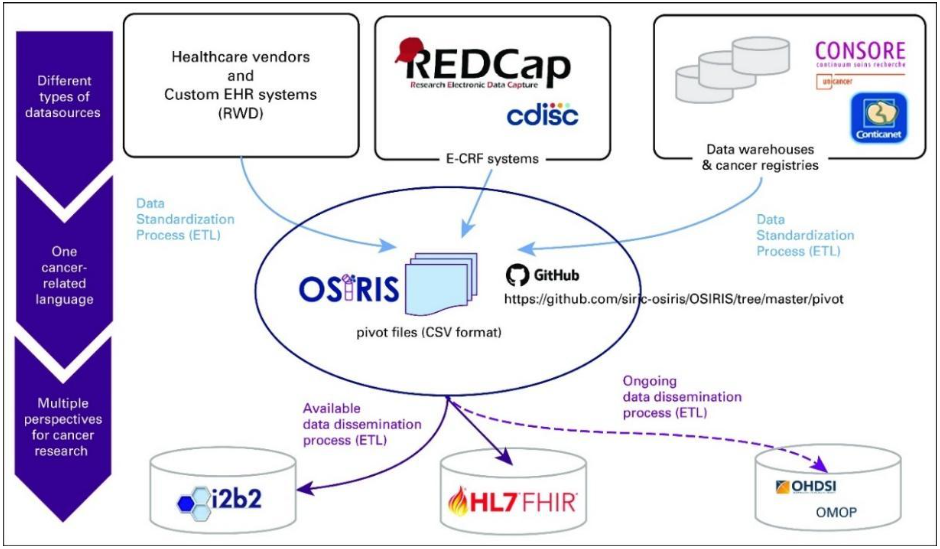
[Figure 5 : Schéma d’intercommunication d'OSIRIS avec des sources de données et avec d’autres standards internationaux, Source : Guérin et al. (2021)
- Communication avec d'autres standards (inter typologies de standards) :
- OSIRIS utilise plusieurs terminologies nationales et internationales, dont la CIM-10 pour les maladies, sa variante dédiée à l'oncologie (CIM-O-3), la CCAM pour les actes médicaux, LOINC pour les concepts génomiques[3:8] (voir Tableau 1 ci-dessous).
# Tableau 1 : Principales terminologies utilisées dans OSIRIS
| Data Domain | National and International Ontologies and Terminologies |
|---|---|
| Patient characteristics | Fast Healthcare Interoperability Resources (FHIR, 3rd edition) Unified Medical Language System (UMLS) WHO classification (performance status) |
| Disease characteristics | International Classification of Disease for Oncology (ICD-O-3, 3rd edition) International Statistical Classification of Diseases and Related Health Problems (ICD, 10th edition) UICC TNM Classification of Malignant Tumors |
| Drug | Anatomical Therapeutic Chemical Classification System (ATC, 5th level) |
| Adverse events | Common Terminology Criteria for Adverse Events (CTCAE, 5th edition) |
| Response evaluation | RECIST, version 1.1 |
| Medical act | Classification of the French Social Security (National Health Service) |
| Genomic concepts | Logical Observation Identifiers Names and Codes (LOINC) HL7 Fast Healthcare Interoperability Resources (HL7 FHIR) |
Source: Guérin et al. (2021)
Flexibilité dans les choix des terminologies :
- Le modèle OSIRIS utilise les terminologies nationales et internationales les plus pertinentes dans chaque domaine pour assurer l'interopérabilité. Lorsqu'il n'existe pas de définition standard, le groupe de travail a créé sa propre terminologie ad hoc (voir la description du travail du groupe technique dans l'élément « Processus de prise de décision sur le standard » en partie 2. Gouvernance, le Tableau 1 ci-dessus pour la liste des principales terminologies utilisées et le Data Supplement n°2 pour la liste exhaustive de l'ensemble des terminologies du modèle[6:4]).
Flexibilité du standard, personnalisation :
- Dans le modèle, conçu pour être modulaire et extensible, il est également possible d'ajouter des données biologiques ou omiques supplémentaires (ex : protéomiques), ainsi que d'autres types de données. Les extensions existantes permettent de traiter les données d'imagerie, les données radiomiques et les données de radiothérapie (voir l'élément « Existence d’extensions certifiées » en partie 5. Utilisation).
Performance :
- Note : 1 / 1 en raison du faible nombre de jointures nécessaires pour réaliser des requêtes. En effet, le niveau de normalisation du modèle étant moyen, le nombre de jointures nécessaires pour chaque requête est restreint.
- L’implémentation native d'OSIRIS utilise le schéma construit autour de la table d'évènement carcinologique (TumorPathologyEvent)[20]. Cette table contient le lien circulaire vers elle-même qui permet de gérer la hiérarchie des événements.
- Par exemple, un événement carcinologique « parent » (ex : cancer du sein) peut être associé à plusieurs autres événements (ex : tumeur primaire, récidive locale, récidive métastatique, voir Figure 6). Dans ce cas-là, l'identifiant de l'événement « parent » sera contenu dans le champ ParentRef et les identifiants des événements « enfants » seront contenus dans les champs InstanceId (voir Figure 7).
- Cependant, cette structure hiérarchique implique d'utiliser soit des requêtes récursives soit une série de requêtes consécutives pour analyser l'évolution de ces événements dans le temps ou d'autres indicateurs qui nécessitent de prendre en compte la relation « parent » - « enfant ».
- Le modèle centré sur les événements d'OSIRIS impose également des restrictions sur la nature de l'organisation des clés étrangères. Au lieu d'une clé simple, il utilise une clé composée de 2 à 4 champs, ce qui impose un nombre minimum de tables impliquées dans l'analyse et, par conséquent, un nombre minimum de jointures.
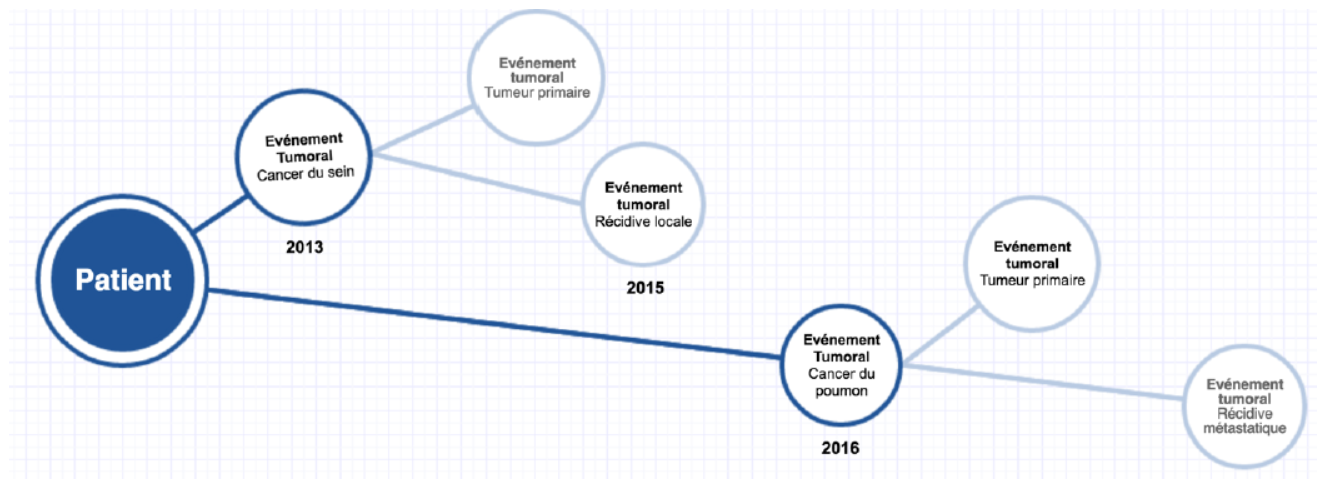
[Figure 6 : Exemple de relations « parent » - « enfants » entre les événements carcinologiques, Source : Description du Modèle de Données Cliniques (version 1.0, 2021)
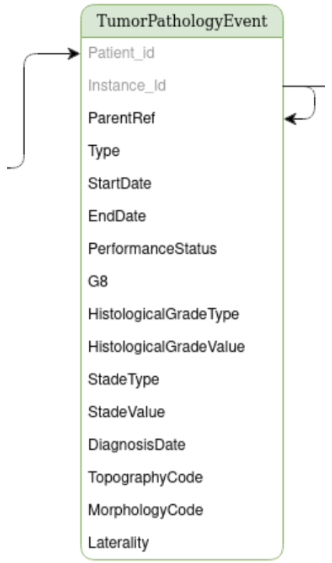
[Figure 7 : Extrait du schéma de données OSIRIS (table TumorPathologyEvent), Source : Schéma de données OSIRIS[20:1]_
Complexité du modèle :
- Note : 0,4 / 1
- Cette note combine plusieurs sous-critères. Elle s’explique par :
- Les nombreuses tables avec des liens entrants et sortants
- Le niveau moyen de normalisation du modèle
- L’absence de tables larges
- Le modèle comporte 18 tables, 213 champs (dont 132 champs cliniques et omiques et 81 champs techniques), et 24 relations.
- Le modèle utilise le type d'organisation des tables « flocon de neige » où certaines tables ne sont pas directement liées à la table d'événement. Cela permet de réduire la redondance d’information en utilisant des relations entre les différentes tables.
- Cependant, toutes les tables ne sont pas normalisées de cette manière : certaines contiennent des informations redondantes au lieu d'utiliser une table de dimension supplémentaire. Par conséquent, le niveau de normalisation n'est pas très élevé.
- Par exemple, les tables Variant, CopyNumber, Fusion et Expression contiennent une partie Validation qui inclut les champs Type, Method et Status pour chaque clé PatientId et Analysis_Ref.
- Pour normaliser ces tables, on pourrait utiliser une table supplémentaire Validation qui contiendrait une clé primaire Validation_Id ainsi que l'ensemble des valeurs pouvant être prises par les champs Type, Method et Status.
- Ainsi, la complexité du modèle OSIRIS est réduite comparativement à un schéma « flocon de neige » complètement normalisé : on observe un équilibre entre l'augmentation de relations entre les tables et la redondance d'information.
Contraintes d'implémentation : OSIRIS n'impose pas de contrainte d'implémentation.
Technologie de stockage et traitement de données et niveau d'adoption de la technologie :
- Le standard ne définit pas la technologie de stockage. Il est possible d’utiliser tous les types de technologies de stockage pour les données structurées (y compris SGBDs, fichiers délimités, etc.).
- À titre d'exemple, dans l'article de Guérin et al. (2021)[3:9], le format CSV est utilisé dans les fichiers pivot pour stocker l'information provenant des établissements de santé dans un format correspondant au schéma OSIRIS avant de la traduire dans un schéma international cible (i2b2, OMOP-CDM, HL7 FHIR, etc.).
Type de technologie de requêtage :
Le standard ne définit pas les technologies de requêtage, elle dépend de l'implémentation.
Neutralité technologique :
OSIRIS est neutre technologiquement. Il définit le schéma de données mais il est indépendant de la technologie utilisée.
Intensité de la perte de données au mapping :
- Le schéma de données OSIRIS a été conçu pour être utilisé comme un modèle commun de l'analyse de la maladie carcinologique, dans une étape intermédiaire entre la lecture des données sources et l'implémentation d'un standard international cible[3:10]. L'utilisation du schéma OSIRIS implique donc deux étapes de mapping (voir l'élément « Capacité de traduction vers un autre standard (intra types de standards) » et la Figure 5 en partie 3. Technique) successives :
- Depuis les systèmes sources de stockage de données dans les établissements de santé vers le langage commun OSIRIS. Au sujet de cette étape de mapping, en date de mai 2023, il n'y a pas eu d'évaluation quantitative de l'intensité de la perte de données.
- Depuis le langage commun OSIRIS vers les standards internationaux d'interopérabilité cibles. Pour cette seconde étape, l'intensité de la perte de données dépend du standard cible choisi et de ses caractéristiques.
- Le schéma de données OSIRIS a été conçu pour être utilisé comme un modèle commun de l'analyse de la maladie carcinologique, dans une étape intermédiaire entre la lecture des données sources et l'implémentation d'un standard international cible[3:10]. L'utilisation du schéma OSIRIS implique donc deux étapes de mapping (voir l'élément « Capacité de traduction vers un autre standard (intra types de standards) » et la Figure 5 en partie 3. Technique) successives :
Compétences techniques et métier nécessaires pour utiliser le standard :
- Profil Data Engineer/Database Administrator pour mettre en place le modèle de données physique pour la technologie de stockage.
- Profil Data Engineer pour développer des flux de données transformant les schémas source vers OSIRIS puis vers le schéma cible (i2b2, OMOP-CDM, etc.)
- Profil informaticien médical ou bio-informaticien pour définir le mapping entre les systèmes source et OSIRIS.
# 4. Valorisation
Accessibilité à des ressources de formation :
En mai 2023, il n'existe pas de ressources de formation (uniquement des ressources de documentation du modèle).
Disponibilité de la documentation scientifique démontrant l'intérêt :
- L'Institut Curie liste l'ensemble de ses projets de recherche en cours, dont deux utilisant le modèle OSIRIS[21] :
- Un projet initié en septembre 2020 (sur environ 110 patients) : étude rétrospective nationale sur les sarcomes des tissus mous chez les enfants, afin de mieux caractériser l'importance du traitement local
- Un projet initié en novembre 2021 (sur environ 200 patients, étude également menée en parallèle au CHU de Bordeaux) : étude des facteurs de modulation de la réponse au traitement pour des patients atteints d'un cancer du poumon métastatique et ayant bénéficié d'une thérapie ciblée sur la base d'une anomalie des gènes EGFR et/ou ALK
- L'Institut Curie liste l'ensemble de ses projets de recherche en cours, dont deux utilisant le modèle OSIRIS[21] :
Adoption du standard :
- Adoptions officielles : En mai 2023, il n'y a pas encore eu d'adoption officielle du modèle OSIRIS.
- Utilisation sur le marché :
- Les institutions membres de l'initiative OSIRIS utilisent déjà le schéma de données (Institut Curie, Institut Bergonié, Centre Léon Bérard, Institut du Cancer de Montpellier, Institut Paoli-Calmettes, Institut Gustave Roussy, CHU de Bordeaux, Hôpital Européen Georges Pompidou, Hôpital Saint Louis, Unicancer)[22]. À terme, l'objectif est de créer un large réseau de bases de données fédérées.
Fournisseurs de service ayant l'expertise en France :
En mai 2023, il n'y a pas de fournisseurs de service ayant l'expertise en France.
Qualité des données :
- Existence d'un label de qualité : Non.
- Outils de vérification de la qualité des données : Non.
# 5. Utilisation
Simplicité d'usage :
- Note : 0,3 / 1
- Cette note combine plusieurs sous-critères. Elle s’explique par :
- L’absence d’accès à des ressources officielles de formation (voir l’élément « Accessibilité à des ressources de formation » en partie 4. Valorisation)
- La lisibilité du schéma par un humain (voir l’élément « Lisible par un humain » en partie 5. Utilisation)
- L’absence d’outils de gestion de la qualité des données (voir l’élément « Qualité des données » en partie 4. Valorisation)
- Le nombre élevé de profils requis pour l’implémentation et l’usage (voir l’élément « Compétences techniques et métier nécessaires pour utiliser le standard » en partie 3. Technique)
- OSIRIS étant un modèle de données théorique qui définit l'ensemble minimal de données à considérer, il est simple à utiliser. L'implémentation du modèle dans un standard international (ex : i2b2, FHIR ou OMOP-CDM) peut cependant s'avérer plus compliquée, en fonction des caractéristiques du standard choisi.
Existence d'une communauté en ligne et degré d'activité : Non.
Outils de mapping : Non.
Outils compatibles : Non.
Décrire les étapes nécessaires pour la standardisation :
- OSIRIS est un langage commun pour l'analyse de la maladie carcinologique. Il est utilisé dans une étape intermédiaire de traduction des données, entre l'import des données sources et la standardisation dans un format international (voir la Figure 5 ci-dessus) :
- Étape 1 : Processus de standardisation des données sources (construction d'un processus ETL)
- Les données sources peuvent exister dans différents formats : EHR, système eCRF (du type REDCap ou cdisc), entrepôts de données (ex : l'entrepôt CONSORE[23] pour les CLCC en France), registre du cancer
- Le processus ETL pour traduire ces données dans le format OSIRIS n'existe pas mais les règles à adopter sont définies dans la spécification et dans les fichiers pivot disponibles sur le GitHub au format CSV[24]. Un processus ETL peut donc être établi dans chaque institution pour traduire les données sources dans le langage OSIRIS
- Étape 2 : Processus de dissémination des données (construction d'un processus ETL)
- Les données traduites dans le langage OSIRIS sont ensuite rendues compatibles avec un ou plusieurs standards internationaux (i2b2, HL7 FHIR, OMOP-CDM), en fonction de l'objectif recherché, de manière à assurer l'interopérabilité.
- Étape 1 : Processus de standardisation des données sources (construction d'un processus ETL)
- OSIRIS est un langage commun pour l'analyse de la maladie carcinologique. Il est utilisé dans une étape intermédiaire de traduction des données, entre l'import des données sources et la standardisation dans un format international (voir la Figure 5 ci-dessus) :
- Existence d’extensions certifiées :
- Il existe plusieurs extensions du modèle OSIRIS : il s'agit des modèles d'imagerie, de radiomique et de radiothérapie (voir Figure 8 ci-dessous pour la radiothérapie), qui permettent de traiter et d'intégrer les données spécifiques concernées[25].
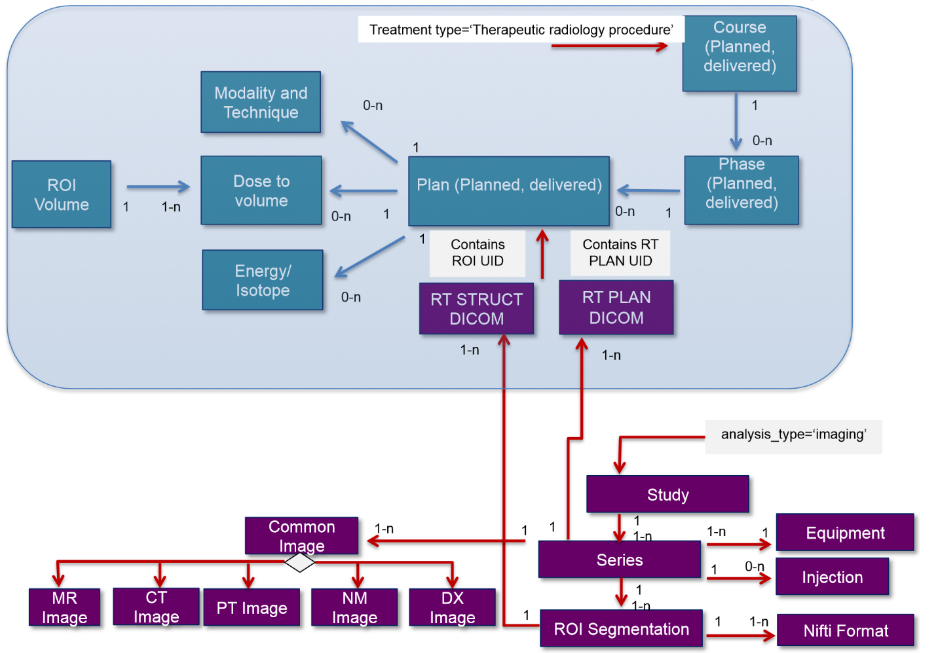
[Figure 8 : Modèle de données de l’extension Radiothérapie, Source : https://github.com/siric-osiris/OSIRIS/tree/MED-OSIRIS/documentation
Bibliothèque de requêtes types : Non.
Lisible par un humain : Oui
- Le modèle OSIRIS se présente sous forme tabulaire et les noms des concepts et des variables sont compréhensibles et renseignent directement sur le contenu. De plus, la succession des événements est également intelligible grâce à la structure longitudinale du schéma.
# Glossaire des acronymes
- ADICAP : Association pour le Développement de l'Informatique en Cytologie et Anatomie Pathologique
- ADN : Acide DésoxyriboNucléique
- ALK : Anaplastic Lymphoma Kinase
- AP-HP : Assistance Publique – Hôpitaux de Paris
- API : Application Programming Interface
- ARN : Acide RiboNucléïque
- CCAM : Classification Commune des Actes Médicaux
- CHU : Centre Hospitalier Universitaire
- CIM : Classification Internationale des Maladies (ICD en anglais)
- CLCC : Centre de Lutte Contre le Cancer
- CNIL : Commission Internationale de l’Informatique et des Libertés
- CSV : Comma Separated Values
- DBA : DataBase Administrator
- DE : Data Element
- DQD : Data Quality Dashboard
- DWH : Data WareHouse
- eCRF : electronic Case Report Form
- EGFR : Epithelial Growth Factor Receptor
- EHR : Electronic Health Record
- ETL : Extract Transform Load
- HL7 FHIR : Heaven Level 7 Fast Health Interoperability Resources
- I2b2 : Informatics for Integrating Biology & the Bedside
- ICD : International Classification of Diseases
- ICD-O-3 : ICD for Oncology, 3rd version
- INCa : Institut National du Cancer
- LOINC : Logical Observation Identifiers Names & Codes
- NGS : Next-generation sequencing
- OHDSI : Observational Health Data Sciences and Informatics
- OMOP-CDM : Observational Medical Outcomes Partnership Common Data Model
- OSIRIS : GrOupe inter-SIRIC sur le paRtage et l’Intégration des donnéeS clinico-biologiques en cancérologie
- RGPD : Règlement Général sur la Protection des Données
- SGBD : Système de Gestion de Base de Données
- SHRINE : Shared Health Research Information Network
- SIRIC : SItes de Recherche Intégrés sur le Cancer
- TNM : Tumor Node Metastases
- TPE : TumorPathologyEvent
Voir : OSIRIS - Une approche « open science » et conceptuelle pour l’analyse de données interopérables en oncologie - Recherche translationnelle (opens new window) ↩︎
Voir l’article de Guérin J, Laizet Y, Le Texier V, Chanas L, Rance B, Koeppel F, Lion F, Gourgou S, Martin AL, Tejeda M, Toulmonde M, Cox S, Hess E, Rousseau-Tsangaris M, Jouhet V, Saintigny P. « OSIRIS: A Minimum Data Set for Data Sharing and Interoperability in Oncology ». JCO Clin Cancer Inform. 2021 Mar : OSIRIS: A Minimum Data Set for Data Sharing and Interoperability in Oncology (opens new window) ↩︎ ↩︎ ↩︎ ↩︎ ↩︎ ↩︎ ↩︎ ↩︎ ↩︎ ↩︎ ↩︎
Il existe deux autres versions d’OSIRIS en cours d'élaboration : OSIRIS RWE et OSIRIS Lung (projet en cours, voir : Le SIRIC BRIO postule pour une 3ème labellisation) (opens new window) ↩︎
Voir : Unicancer (opens new window) ↩︎
Voir la documentation des Data Supplements 1, 2 et 3 pour une description précise des éléments de données et de l’ensemble des valeurs qu’ils peuvent prendre : https://ascopubs.org/doi/suppl/10.1200/CCI.20.00094 ↩︎ ↩︎ ↩︎ ↩︎ ↩︎
Voir la Description du Modèle de Données Cliniques (version 1.0, 2021) : https://github.com/siric-osiris/OSIRIS/blob/master/documentation/OSIRIS_Sp%C3%A9cifications_Mod%C3%A8le_Donn%C3%A9es_Cliniques_TC_v1.0_(DRAFT).pdf ↩︎ ↩︎
Voir le GitHub : https://github.com/siric-osiris/OSIRIS/tree/master/documentation ↩︎
Voir : https://github.com/siric-osiris/OSIRIS/wiki/DataElementConcept ↩︎
Voir le troisième document Data Supplement qui compare notamment l’existence de licences associées à OSIRIS mCODE et OMOP-CDM : https://ascopubs.org/doi/suppl/10.1200/CCI.20.00094. Pour OSIRIS, il est indiqué : « Licensing : None » ↩︎
Principalement l'Institut Curie, l'Institut Bergonié, le Centre Léon Bérard, l'Institut Gustave Roussy, l'Institut du cancer de Montpellier, l'Institut Paoli-Calmettes. ↩︎
Principalement le CHU de Bordeaux, l’Hôpital Européen Georges Pompidou et l’Hôpital Saint-Louis de l'Assistance Publique-Hôpitaux de Paris (AP-HP). ↩︎
CARPEM, CURAMUS, Curie, EpiCURE, ILIAD, InSiTu, LYriCAN, Montpellier Cancer : Les SIRIC - Recherche translationnelle (opens new window) ↩︎
Voir : https://fhir.arkhn.com/osiris/ ↩︎
Voir la procédure de contribution : https://github.com/siric-osiris/OSIRIS/wiki/How-to-contribute ↩︎ ↩︎
Voir la page : Issues · InstitutNationalduCancer/OSIRIS · GitHub (opens new window) ↩︎
Voir la liste des versions : https://github.com/siric-osiris/OSIRIS/releases ↩︎
Voir le guide d’implémentation FHIR d’OSIRIS : https://fhir.arkhn.com/osiris/ ↩︎
Voir le guide d’installation Docker pour déployer l’instance i2b2/SHRINE : GitHub - CARPEM/SHRINEDocker: A Docker version of SHRINE/i2b2 (opens new window) ↩︎
Voir : https://github.com/siric-osiris/OSIRIS/blob/master/documentation/MPD_OSIRIS_model_v1.1.05.png ↩︎ ↩︎
Voir : https://curie.fr/sites/default/files/medias/documents/2023-04/LISTING%20NOS%20PROJETS%20DE%20RECHERCHE%20V54.pdf ↩︎
Voir : OSIRIS : a national data sharing project - www.en.ecancer.fr (opens new window) ↩︎
Voir la documentation sur l’entrepôt CONSORE : Programme Consore : Structuration données médicales et Big Data du cancer - Unicancer (opens new window) ↩︎
Voir : https://github.com/siric-osiris/OSIRIS/tree/master/pivot ↩︎
Voir la liste des extensions : https://github.com/siric-osiris/OSIRIS/tree/MED-OSIRIS/documentation ↩︎