# SCALPEL, une chaîne de traitement pour transformer le SNDS
Crédits
Cette fiche a été initialement rédigée par Matthieu Doutreligne, DREES en s'appuyant sur la documentation de SCALPEL (opens new window). Merci à Arnaud Lecoules (DREES) pour les schémas d'aplatissement.
À savoir : Des références peuvent être données à la fin de la fiche. La dernière date de modification se situe tout en bas à droite.
Le SNDS est issu de la fusion de différentes bases de données médico-administratives (SNIIRAM, PMSI, CépiDC, ...). La plupart de ces sources ont pour but premier la facturation. Malgré la richesse de ces données, la structure brute du SNDS est donc hétérogène et peu adaptée à l'analyse.
La quasi totalité des études portant sur le SNDS commence par appliquer des filtres de qualité puis par réunir les informations d'intérêts dans les différents produits du SNDS. Ces démarches sont coûteuses en temps, en ressources de calculs et peuvent être implémentées différemment d'une étude à l'autre.
SCALPEL est une chaîne de traitement qui vise à
- améliorer la comparativité et la reproductibilité des études en normalisant le nettoyage des données,
- accélerer les traitements en s'appuyant sur des technologies Big Data à l'état de l'art calcul distribué (Spark) (opens new window),
- extraire à partir du SNDS des informations médicales de manière structurée et testée à l'aide de tests unitaires.
Cette chaîne de traitement a été initiée dans le cadre du partenariat CNAM-Polytechnique dans l'équipe d'Emmanuel Bacry, Stéphane Gaiffas et Youcef Sébiat. Elle a donné lieu à des publications scientifiques, SCALPEL-3, Bacry et al. 2019[1], ZiMM, Bacry et al., 2019[2]. Les codes sources sont disponibles sur le github du partenariat : SCALPEL-Flattening (opens new window) et SCALPEL-Extraction (opens new window).
Ces deux projets (en langage informatique scala) sont utilisés pour transformer des extractions CNAM du SNDS en des vues et des formats plus accessibles pour l'analyse et la recherche observationnelle.
SCALPEL-Flattening : Une première étape simplifie la structure en aplatissant le SNDS afin de réduire la structure en étoile et éviter les jointures coûteuses.
SCALPEL-Extraction : Une seconde étape intègre les filtres usuels de qualité de la donnée du SNDS, et des logiques d'extraction de tables événementielles permettant une analyse simplifiée des trajectoires patients.
# SCALPEL-Flattening (opens new window), l'aplatissement du SNDS
Cette première étape transforme les tables d'origine .csv en tables parquet. Ce format de fichiers distribués orientés colonne permet de tirer partie du framework de calcul distribué spark. Une seconde étape effectue des jointures des tables affinées autour des tables centrales de chaque produit. Mis à part les clés de jointure, il n'y a pas de connaissance métier dans cette partie.
Actuellement, les tables obtenues sont : DCIR, MCO, MCO_CE, SSR, SSR_CE, HAD, RIP, RIP_3A. L'extension _CE désigne les consultations externes. Nous avons construit trois types d'aplatissement adaptés aux structures des différents produits du SNDS : le plus simple est utilisé pour obtenir la base de données DCIR (cf. Fig. 1) et les deux autres pour les différentes bases de données PMSI. Les différentes méthodes sont détaillées sur le github du projet (opens new window) en anglais.
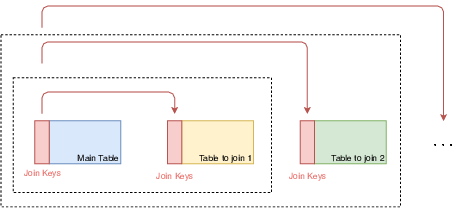
Fig. 1, Logique de jointure pour le DCIR
# SCALPEL-Extraction (opens new window), extraire des tables d'événements
# Le format d'arrivée
La deuxième partie de la chaîne de traitement transforme les données du SNDS afin de leur donner une forme simple pour l'analyse, notamment pour les études orientées patient et se focalisant sur la temporalité (ex: parcours de soins).
Le résultat recherché est un format condensé où chaque contact entre un patient et le système de soin (un contact étant désigné par le terme événement) peut facilement être mis en relation avec un autre. Le tableau suivant donne une idée de ce format d'arrivée :
| patientID | category | groupID | value | weight | start | end |
|---|---|---|---|---|---|---|
| 0000001 | mco_ccam_act | eta_num__rsa_num | GLQPOO2 | 0.0 | 2016-12-01 | null |
| 0000002 | drug | 9_cles_jointures | A10BX06 | 0.0 | 2016-05-24 | null |
La colonne value correspond au code médical correspond à l'événément extrait (ex. GLQP002 est l'acte CCAM de mesure de la capacité vitale lente, A10BX06 est le code ATC du Benfluorex). Le groupID recense de manière unique le contact avec le système de soin. La colonne weight désigne une valeur numérique s'il y a lieu (ex. nombre de comprimés).
Une description fine des différents composants de l'extraction est disponible sur le github du projet : composant Event (opens new window), composant Extractor (opens new window), composant Transformer (opens new window). Cette documentation permet à un utilisateur novice de la pipeline d'étendre le code d'extraction pour de nouvelles études.
# Les filtres de qualité de SCALPEL-Extraction
Les sources du SNDS utilisées pour l'extraction sont les données brutes d'une extraction CNAM aplaties par produits par l'étape une de SCALPEL. Elles doivent donc être nettoyées avant toute extraction d'événements.
Nous utilisons quelques règles classiques de qualité de la donnée dans le SNDS. Selon les produits concernés, on peut se référer aux fiches spécialisées sur ce site de documentation.
DCIR :
DPN_QLF ≠ "71", supprime les lignes pour remontée d'information : ces lignes correspondent à des prestations effectuées lors d'un séjour ou d'une consultation ambulatoire et envoyées à titre indicatif,BSE_PRS_NAT ≠ "0", supprime les lignes pour lesquelles la nature de la prestation est sans objet,
PMSI :
Pour tous les produits du PMSI (voir détails ici) :
ETA_NUM $\not in$ finess_doublon: supprime les FINESS remontant en doublon pour les hôpitaux APHP, HCL et APHM (les informations pour ces établissements remontent également par le FINESS juridique),MCO :
SEJ_TYP = $\empty$ OR SEJ_TYP ≠ "B" OR (GRG_GHM like "28%" AND GRG_GHM $\notin$ {"28Z14Z", "28Z15Z", "28Z16Z"}), SEJ_TYP == B correspond aux prestations inter-établissements (PIE) dont les informations sont des doublons sauf pour lesGRG_GHMlistés correspondants aux dialyses et radiothérapies.GRG_GHM ≠ "14Z08Z", supprime les séjours hospitaliers en IVG (pour des questions légales : critère à vérifier)GHS_NUM ≠ "9999", supprime les séjours hospitaliers non remboursés,{SEJ_RET, FHO_RET, PMS_RET, DAT_RET, NIR_RET, SEX_RET} = "0", supprime les lignes pour lesquelles l'identifiant ou le sexe du patient sont en erreur,GRG_GHM not like "90%", supprime les codes GHM corrompus,
MCO_CE :
- {NAI_RET, IAS_RET, ENT_DAT_RET, NIR_RET, SEX_RET} = "0", supprime les lignes pour lesquels l'identifiant patient ou le sexe sont en erreur,
SSR :
{NIR_RET, SEJ_RET, FHO_RET, PMS_RET, DAT_RET} = "0", supprime les lignes pour lesquelles l'identifiant du séjour, la date ou l'identifiant du patient sont en erreur,GRG_GME \not like "90%", supprime les codes GHM corrompus,
SSR_CE :
{NIR_RET, NAI_RET, SEX_RET, IAS_RET, ENT_DAT_RET} = "0", supprime les lignes pour lesquelles l'identifiant du séjour, la date ou l'identifiant du patient sont en erreur,
HAD :
{NIR_RET, SEJ_RET, FHO_RET, PMS_RET, DAT_RET} = "0", supprime les lignes pour lesquelles l'identifiant du séjour, la date ou l'identifiant du patient sont en erreur.
E. Bacry et al., « SCALPEL3: a scalable open-source library for healthcare claims databases », arXiv:1910.07045 (opens new window) [cs], oct. 2019. ↩︎
E. Bacry, S. Gaïffas, A. Kabeshova, et Y. Yu, «ZiMM: a deep learning model for long term adverse events with non-clinical claims data », arXiv:1911.05346 (opens new window) [cs, stat], nov. 2019. ↩︎